在生物技术的前沿领域,AI 正在掀起一场蛋白质研究的革命。AI 帮助科学家以前所未有的精度预测蛋白质结构和功能、从头设计新型蛋白质、解开生命分子层面的奥秘。蛋白质生产技术的进步,如AlphaFold 和人工智能驱动技术,不仅改写了蛋白质研究的规则,也为从药物开发到疾病诊断和合成生物学等各个领域带来了许多可能性。
人工智能在蛋白质结构预测中的作用
蛋白质研究一直是生物技术领域中的核心难题,其复杂性源于蛋白质多样的三维结构和动态特性。长期以来,科学家们一直使用X射线晶体学、核磁共振(NMR)和冷冻电子显微镜等传统工具来解析蛋白质结构。然而,这些方法往往成本高昂、耗时,并对特定蛋白质类型的解析存在局限性。这些传统技术在应对蛋白质的动态性和环境依赖性方面也有较大挑战,导致实际解析的结构可能无法准确反映蛋白质在生理条件下的状态。科学家们亟需新的工具,能够更快速、高效、低成本地完成复杂蛋白质结构的预测与解析,以满足现代生命科学研究的需求。
AI如何突破蛋白质研究困境?
随着计算机技术和算法的不断进步,科学家们开始探索使用人工智能和机器学习方法来解决蛋白质结构预测问题。
2020年,DeepMind 团队推出了突破性工具 AlphaFold,并经历了不断的发展和完善。该模型利用深度学习精确预测蛋白质三维结构,显著提升了蛋白质结构预测的准确性和速度,为生物学研究带来了革命性进展。AlphaFold 使用深度学习模型,通过分析多序列比对 (MSA) 中的进化协变数据,预测氨基酸残基之间的距离,从而揭示蛋白质结构的空间构造。。神经网络会预测一个 “距离图 ”或残基-残基距离的概率图,为折叠过程提供指导。利用这些距离信息,该模型会进行优化(如梯度下降),以确定蛋白质的最终三维结构。
2020年,AlphaFold 2.0 在蛋白质结构预测竞赛 CASP14 上一鸣惊人,获得了98.5%的蛋白质结构预测率。作为 Alphafold 的升级版本,它能在几分钟内预测出典型蛋白质的结构,精度通常在 1 Å 以内,接近碳原子宽度(约 1.4 Å)。这种精确度代表了计算生物学的重大飞跃,为了解蛋白质折叠的复杂性提供了强有力的工具。
AlphaFold 2.0 引入了一种名为 Evoformer 的先进神经网络架构。该架构利用进化和空间关系处理多序列比对(MSA)和成对残基信息。通过三角更新和注意力机制,帮助模型捕捉远程依赖性和空间关系,这对准确预测蛋白质折叠至关重要。它还包括一个结构模块,直接预测三维原子坐标,并采用循环机制进行迭代优化。通过端到端训练和独特的自蒸馏过程,AlphaFold 2达到了接近实验的精度,能够预测以前具有挑战性的复杂和新型结构。
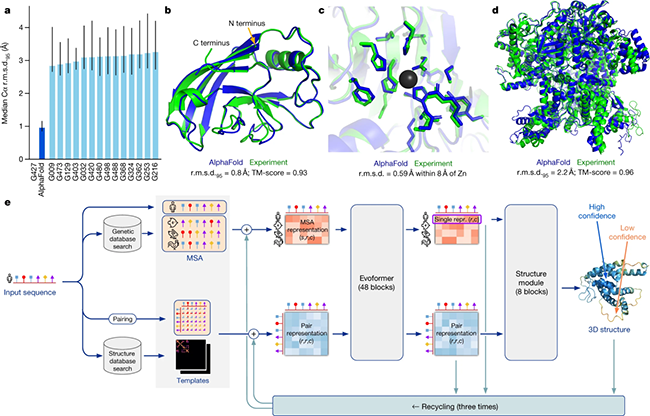
AlphaFold 2.0 模型架构
2021年, David Baker的团队推出了革命性工具 RoseTTAFold。这一开源蛋白质结构预测工具采用了独特的三轨网络架构,能够同时处理序列、距离和坐标信息,使得预测的精准度和速度显著提高。
• 序列轨道:处理氨基酸序列信息。
• 距离轨道:处理氨基酸对之间相互作用的信息。
• 坐标轨道:处理局部结构特征,如二级结构和溶剂可及性。
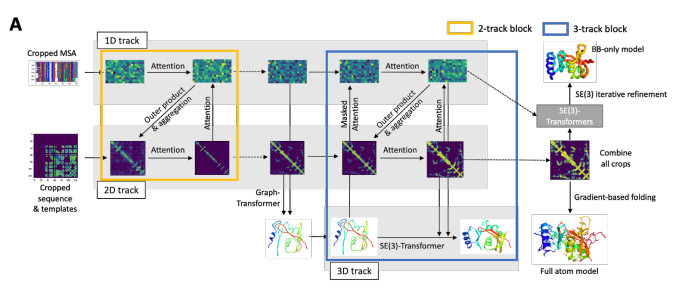
RoseTTAFold 的三维轨道架构
该模型在不同通道中同时处理序列、距离和坐标信息,通过通道间的信息不断交换来迭代完善蛋白质结构。RoseTTAFold 的设计实现了一种多任务学习方法,可以同时优化多个相关任务,如距离图预测、角度图预测和接触图预测,有助于提高整体预测精度。
人工智能在蛋白质功能预测中的作用
蛋白质的功能是在基因本体(GO)中被定义的,其依据分子功能(MFO)、在生物过程里的作用(BPO)以及在细胞成分中的位置(CCO)来对蛋白质实施分类。借助对同源蛋白质的注释,诸如UniProtKB/Swiss - Prot这类数据库,为数千种生物以及超过55万种蛋白质提供了经过整理的GO数据。
然而,数据库中大部分蛋白质缺少功能注释,现有的注释大多源于耗时的实验。基于AI的预测方法,融合了氨基酸序列、结构信息以及蛋白质 - 蛋白质相互作用(PPI)网络,使得蛋白质功能预测更加高效,显著提高了预测准确性和速度。这为填补蛋白质功能注释的空白提供了可扩展的解决方案。。通过运用深度学习和文献知识,这些工具能够更高效且更精准地进行功能预测,从而加深我们对蛋白质在健康和疾病中所起作用的理解。
DeepGO 是第一个基于深度学习的预测模型,它通过将深度学习应用于蛋白质序列和相互作用数据来预测蛋白质功能。该模型以蛋白质的氨基酸序列作为输入,使用卷积神经网络(CNNs)从中提取特征。DeepGO 还结合了蛋白质-蛋白质相互作用(PPI)网络,使模型能够利用蛋白质之间的功能关系。通过这种方法,DeepGO 分配基因本体(GO)术语,根据分子功能、生物过程和细胞成分对蛋白质功能进行分类,这对全面的蛋白质功能预测很有效果。
DeepGO-SE 是一种用于蛋白质功能预测的高级模型,它通过一种称为近似语义蕴含的过程,将基因本体(GO) 中的知识纳入其中。该模型的运行分为三个关键步骤:
1. 构建近似模型:利用GO的公理和蛋白质功能断言创建近似模型,其中ELEmbeddings以几何表示法捕捉GO 中的语义关系。
2. 蛋白质嵌入和优化:蛋白质序列用预训练的ESM2模型的嵌入表示。然后在近似模型中对这些嵌入进行定位,以最大限度地提高“蛋白质具有C功能 ”这样的语句的可能性,从而指导精确的功能预测。
3. 多模型聚合:重复这个过程以生成多个模型,最终预测基于所有模型中都成立的真值,有效地捕捉蕴含关系。
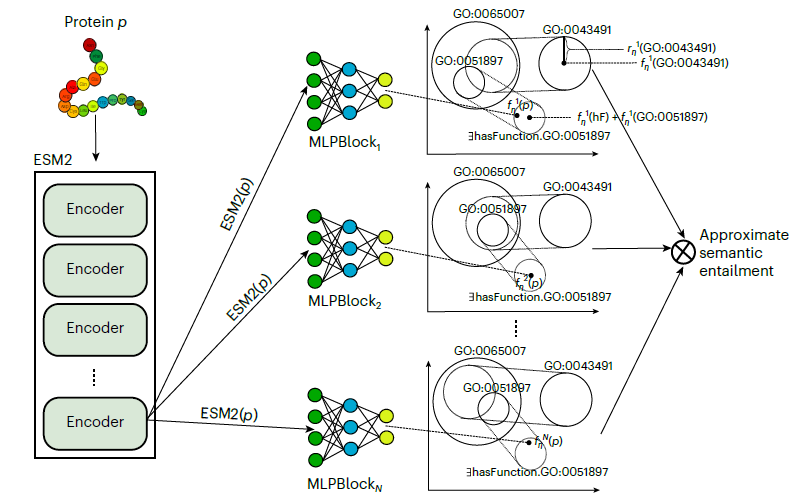
DeepGO-SE模型
人工智能在蛋白质设计中的应用
蛋白质设计已经有了显著的发展,从最初通过PCR的诱变引入特定突变来调控蛋白质结构,到应用先进计算方法构建具有所需特性的新型蛋白质。如今,随着结构生物学、计算建模的快速发展,AI蛋白质设计比以往任何时候都更加精确,也更容易获得。AI驱动的设计技术既能优化蛋白质,增强蛋白质的天然功能(如亲和力和稳定性),还具备从头构建全新蛋白质的能力,以实现特定的功能、结构和应用,为药物发现、工业酶工程等领域带来前所未有的创新可能性。
ProteinMPNN 是一种用于高效蛋白质序列设计的深度学习模型,它绕过了传统基于物理的方法(如Rosetta)的计算需求。通过直接从结构数据中学习并编码空间关系, ProteinMPNN 能准确预测折叠成目标结构的序列。该模型在设计复杂的组装(如四面体纳米粒子)和保持结合亲和力(即使发生点突变)方面表现出色。该模型能够创建各种类型的蛋白质,包括单体、组装体和纳米颗粒,是提高蛋白质溶解度、稳定性和功能性的强大工具。
RFdiffusion 采用创新方法,利用扩散模型,通过迭代去噪,将蛋白质骨架从最初的噪音细化为现实结构。通过结合特定的结构基序,它创造出多样化、复杂的蛋白质,以满足特定需求,如对称组装体和功能基序。RFdiffusion在生成新型拓扑结构方面的精确性使其能够应用于前沿领域,从治疗支架到复杂结构设计。
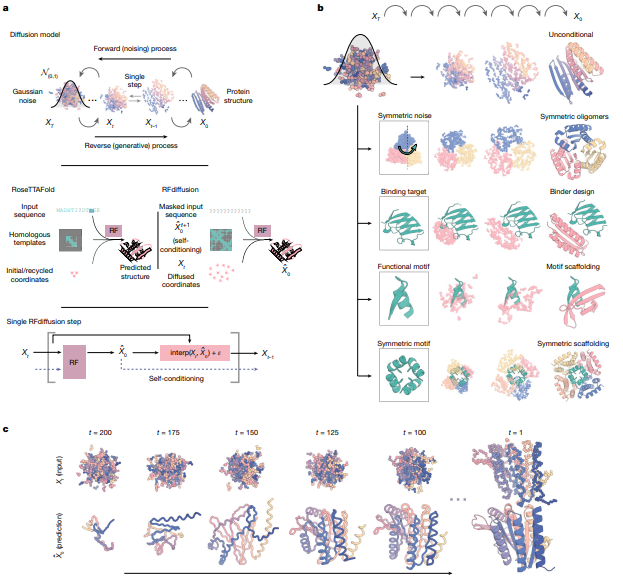
利用RFdiffusion设计蛋白质
ProGen 借鉴NLP的语言模型技术,生成反映进化模式和生化特性的蛋白质序列,从而能够创建具有特定功能的序列。ProGen使用转换器来模拟驱动稳定性和功能性的序列特征,使其高度适用于各种应用,从酶设计到生成具有特定结合亲和力的序列。
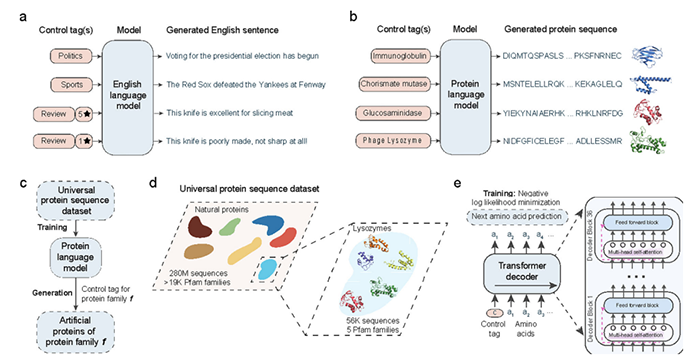
利用条件语言建模生成人工蛋白质
AlphaProteo 专注于创建高亲和力蛋白质结合剂,利用结构引导序列生成技术开发针对精确蛋白质位点的结合剂。通过将生成模型与高级过滤器相结合。AlphaProteo在生产针对具有挑战性的目标(如病毒和癌症相关蛋白质)的结合剂方面表现出色,在许多情况下实现了亚纳摩尔亲和力。它能够简化粘合剂设计,减少优化次数,为治疗开发开辟了新的可能性。
AlphaProteo 在测试的七种目标蛋白质上都有较高的实验成功率。在湿实验室测试中,9% 到 88% 的候选分子成功结合,比其他方法高 5 到 100 倍;比现有最佳方法的结合亲和力高 3 到 300 倍。
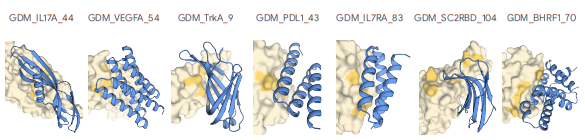
从AI蛋白设计到湿实验验证
研究人员现在能够直接从序列和结构数据中生成针对特定功能(如结合亲和力、稳定性和催化活性)优化的蛋白质。然而,将这些计算设计转化为功能性、可靠的蛋白质需要严格的湿实验验证,以确保结合亲和力、稳定性和生物活性等特性。在湿实验验证过程中,科学家面临许多挑战,复杂蛋白质的结构和性质可能导致其在表达过程中出现折叠异常或形成无活性的聚集体,极大地降低表达效率。湿实验的数据反馈至关重要,可用于进一步优化AI模型,从而提高模型对未来设计的精准度和有效性。
必威生物为研究人员提供了一站式解决方案,简化了从数字序列到经过实验验证的蛋白质产品的过程。与我们合作,您只需提供蛋白质序列,我们会处理从密码子优化和基因合成到表达系统选择、蛋白质纯化和功能验证的每一个细节。
- 密码子优化:我们的NG Codon技术提高表达,针对您的特定表达系统进行了优化。
- 基因合成与克隆:高保真基因合成并克隆到任何指定的载体中。
- 表达系统筛选:可使用细菌、酵母、昆虫和哺乳动物宿主进行定制表达。
- 重组抗体表达:通过整合抗体基因序列、从头抗体设计、抗体人化、抗体基因合成、重组抗体表达、单克隆抗体制备和多克隆抗体制备,涵盖抗体发现的所有阶段。
- 大规模蛋白质生产:灵活的生产规格,从微克到克,以支持任何规模的项目。
- 质量与功能验证:全面的测试确保最终蛋白质或抗体产品的可靠性、活性和功能性。
全球AI蛋白质公司

部分公司,联系我们申请完整版本
AI在蛋白质科学中的应用多样性已经在重塑药物发现、精准医学和合成生物学。由David Baker等先驱孵化的公司正在使用深度学习来解决关键的生物学挑战,包括药物开发、多肽设计、小分子结合蛋白质工程和新型材料合成。随着AI加速和增强蛋白质设计的能力得到证明,其在生物技术中的作用预计将不断增长,为定制疗法和创新生物材料开辟新的可能性,并推动生命科学领域的边界不断拓展。
References
[1] Jumper, John, et al. "Highly accurate protein structure prediction with AlphaFold." nature 596.7873 (2021): 583-589.
[2] Baek, Minkyung, et al. "Accurate prediction of protein structures and interactions using a three-track neural network." Science 373.6557 (2021): 871-876.
[3] Madani, Ali, et al. "Large language models generate functional protein sequences across diverse families." Nature Biotechnology 41.8 (2023): 1099-1106.
[4] Senior, Andrew W., et al. "Improved protein structure prediction using potentials from deep learning." Nature 577.7792 (2020): 706-710.
[5] Zambaldi, Vinicius, et al. "De novo design of high-affinity protein binders with AlphaProteo." arXiv preprint arXiv:2409.08022 (2024).
[6] Kulmanov, Maxat, et al. "Protein function prediction as approximate semantic entailment." Nature Machine Intelligence 6.2 (2024): 220-228.
[7] Watson, Joseph L., et al. "De novo design of protein structure and function with RFdiffusion." Nature 620.7976 (2023): 1089-1100.
 Syno®C 引物合成
Syno®C 引物合成 RNA合成
RNA合成 mRNA合成
mRNA合成 Syno®GS 基因合成
Syno®GS 基因合成 载体构建
载体构建 高通量及DNA文库构建
高通量及DNA文库构建 CRISPR基因编辑平台
CRISPR基因编辑平台 病毒包装
病毒包装 基因测序及分析
基因测序及分析 重组蛋白表达平台
重组蛋白表达平台 抗体工程平台
抗体工程平台 多肽服务
多肽服务 生物信息学分析与设计
生物信息学分析与设计 CRISPR文库
CRISPR文库 ProXpress蛋白快速检测
ProXpress蛋白快速检测 CRISPR 质粒
CRISPR 质粒





















